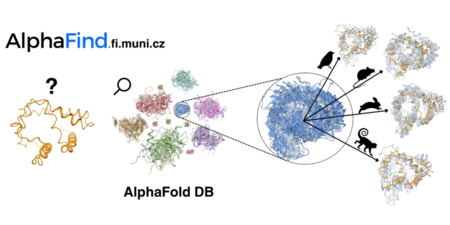
AlphaFind: nový kompas ve světě proteinů
Vědecký tým z Masarykovy univerzity (MU) představil novinku v oblasti bioinformatiky - AlphaFind, webový nástroj určený k snadnějšímu vyhledávání proteinů v databázi AlphaFold (AlphaFold DB). Slibuje rychlejší orientaci v rozsáhlých datech, možnost 3D vizualizací i všeobecnou dostupnost a přívětivost všem uživatelům.
Proteiny neboli bílkoviny jsou základními stavebními kameny našich buněk a hrají klíčovou roli v jejich fungování. Jednotlivé proteiny se od sebe navzájem liší především pořadím aminokyselin, které obsahují. AlphaFold DB pracuje s více než 200 miliony vzorků bílkovin a je tak pro vědce jako knihovna, ze které si půjčují konkrétní proteiny pro svůj výzkum. Na základě sekvence aminokyselin dokáže databáze s využitím umělé inteligence předpovídat 3D strukturu jednotlivých proteinů, což pomáhá porozumět jejich biologickým funkcím a rozvíjet například účinné metody prevence či léčby nemocí. Obsáhlost celé databáze však klade nároky na zpracování velkého množství dat při vyhledávání konkrétních vzorků. A zde přichází ke slovu nový pomocník z dílny Masarykovy univerzity - AlphaFind.
„Hlavním cílem AlphaFind je poskytnout rychlou, škálovatelnou a uživatelsky přívětivou platformu pro vyhledávání a porovnávání proteinových struktur v rámci AlphaFold DB. Na rozdíl od tradičních nástrojů, které se často potýkají s velkými soubory dat, se AlphaFind zaměřuje na terciární struktury proteinů a využívá pokročilé modely strojového učení k efektivní identifikaci kandidátních proteinů a následnému podrobnému porovnání jejich 3D struktur,” vysvětluje Vlastislav Dohnal z Katedry strojového učení a zpracování dat Fakulty informatiky MUl, jeden z autorů nástroje.
AlphaFind slibuje hned několik výhod. Díky novému přístupu k indexování, který výrazně snižuje velikost dat z přibližně 23 terabajtů na 20 gigabajtů, efektivně zpracovává celou databázi. Vyhledávač poskytuje rychlé výsledky vyhledávání, obvykle vrací 50 nejpodobnějších struktur přibližně za 7 sekund. Samotná webová aplikace je navržena tak, aby se snadno používala, nevyžaduje registraci a podporuje různé vstupní formáty, například UniProt ID, PDB ID a genové symboly. Obsahuje také nástroje pro 3D vizualizaci superpozic proteinových struktur, což umožňuje okamžitě analyzovat strukturní podobnosti.
Za vývojem AlphaFind stojí hned 4 pracoviště MU. Jádro aplikace a její infrastrukturní zázemí vyvinulo centrum CERIT-SC Ústavu výpočetní techniky, které zároveň celou spolupráci koordinovalo. Mechanismus vyhledávání podobnosti struktur je postaven na výzkumu skupiny Intelligent Systems for Complex Data Fakulty informatiky. Výzkumníci z Biological Data Management and Analysis Core Facility z CEITEC a Národního centra pro výzkum biomolekul Přírodovědecké fakulty pak vyhodnocovaly kvalitu a použitelnost z pohledu strukturní biologie. AlphaFind je tak zářným příkladem fungující synergie v rámci MU.
Celý tým nedávno publikoval svoje výsledky v časopise Nucleic Acids Research, kde nový nástroj představil a nabídl k širšímu využití celé vědecké komunitě. Celý článek najdete zde: https://doi.org/10.1093/nar/gkae397.
AlphaFind je k dispozici online a zdarma na adrese https://alphafind.fi.muni.cz. Uživatelská příručka a zdrojový kód jsou k dispozici na GitHubu.
Autorka: Marta Vrlová, Oddělení vnějších vztahů a spolupráce s partnery FI MU
AlphaFold
Již od 70. let 20. století vzniká Protein Data Bank – databáze existujících proteinů, která je vědecké komunitě k dispozici pro související výzkum. Na jejím základě pak v roce 2018 postavila společnost DeepMind nástroj AlphaFold, která dnes obsahuje přes 200 miliony vzorků bílkovin, převážně lidského původu, ale i dalších organismů významných pro genetický výzkum. Za využití AI pak podle sekvence aminokyselin dokáže databáze předpovídat 3D strukturu jednotlivých druhů proteinů, což je klíčové pro porozumění jejich biologickým funkcím. Tyto zjednodušené modely bílkovin pak pomáhají například při vývoji nových léků, kdy zkoumáme vliv těchto přípravků na jednotlivé části proteinů a můžeme pak léčbu efektivněji zacílit. 8. května 2024 vývojáři představili novou verzi AlphaFold3, která dokáže modelovat i interakci proteinů s ostatními molekulami, což opět významně posouvá výzkumné možnosti.
Intelligent Systems for Complex Data Research Group
Jsme výzkumný tým z Masarykovy univerzity v Brně se specializací na komplexní analýzu dat. V rámci laboratoře Laboratoř datově orientovaných systémů a aplikací na FI MU a v úzké spolupráci s centrem CERIT-SC se snažíme objevovat zákonitosti v obrovském množství komplexních dat. Řešíme jedinečné výzvy od zkoumání vzorů v obrazech až po složitou analýzu komplexních biologických struktur, jako jsou proteiny. Naší ambicí je nově definovat hranice efektivního a účinného zpracování rozsáhlých souborů dat s využitím našich znalostí v oblasti strojového učení, dolování dat a technik shlukování.